新冠病毒引发的传染性肺炎COVID-19已经蔓延全球,世界卫生组织(WHO)将其列为全球性流行病(Pandemic),是需要全人类共同努力战胜的传染病。虽然在中国、韩国、日本等部分国家,新冠肺炎的传播已经得到一定程度的遏制,但是由于还没有特效药和疫苗,抗疫局势仍然不容乐观。与17年前的非典肺炎(SARS)类似,新冠肺炎是由一种冠状病毒引发的,其基因组与SARS病毒有较高相似度,因此,这个病毒被命名为SARS-CoV-2。研究结果表明,SARS-CoV和SARS-CoV-2病毒入侵细胞的主要受体蛋白都是人体细胞的血管紧张素转换酶2(简称ACE2)。病毒表面的棘突蛋白与ACE2紧密结合之后,再进一步融合到人体细胞内,实现入侵和复制。因此,以任何方式切断或者干扰棘突蛋白与ACE2的相互作用对于控制病毒入侵至关重要。
北京计算科学研究中心(刘海广课题组)、北京大学BIOPIC(苏晓东实验室)、四川大学华西医院(雷剑课题组)及中科院微生物所(齐建勋)等单位的科研人员利用超级计算机研究了SARS-CoV-2的棘突蛋白与人体ACE2的结合模式和动态相互作用,并用同样的方法研究了氨基酸突变的ACE2蛋白与棘突蛋白的相互作用,旨在全面理解棘突蛋白与人体ACE2的动态结合过程,为相关阻断其结合的药物设计打下基础,也为理解其他哺乳动物的ACE2与新冠病毒的结合开辟了方向。结果表明棘突蛋白的受体结合区(RBD)与人体的ACE2紧密结合,两种SARS-CoV和SARS-CoV-2病毒棘突蛋白结合能力类似,而如果将ACE2的几个重要氨基酸突变为大鼠的ACE2序列,则结合模式和紧密程度都受到较大影响,该结果已经发布到预印版BioRxiv(doi: https://doi.org/10.1101/2020.03.24.005561)。
该项目从1月底疫情刚爆发时就积极开展,由于当时复合物的晶体结构还没有解析出来,研究人员利用同源建模的方法根据SARS-CoV棘突蛋白结构预测了SARS-CoV-2的RBD结构并构造了RBD与人体ACE2的复合物结构。以此为基础,研究人员利用北京超级云计算中心和北京计算科学研究中心的超级计算机,对棘突蛋白与人体ACE2的复合物进行了大量的全原子动力学模拟(MD)。在项目的进行过程中,该复合物分子的晶体结构被我国两个实验组独立快速解析出来并及时进行了发表前的共享。通过与晶体结构对比,研究团队发现预测的结构准确度非常高,尤其是在棘突蛋白与ACE2结合部位几乎完全一致,证明预测的结构具有很高的可靠性(图a, b)。为了进一步对比,研究人员又以实验测定的结构为基础做了对照模拟。在三条野生型ACE2与棘突蛋白RBD的复合物的模拟中,尽管ACE2和棘突蛋白各自展示出一定的结构变化,但是棘突蛋白的结合域与ACE2都始终保持紧密结合,紧密接触的氨基酸平均保持在33对(图c)。ACE2的结构变化对应其生理功能,围绕着它的酶催化中心有开合的运动。棘突蛋白结合域的结构变化则主要表现在一些柔性的连接区域。当把人体ACE2的一些氨基酸突变为大鼠(Rat)的ACE2之后,棘突蛋白与突变的ACE2结合就变得不稳定。第一种突变围绕着ACE2的N端第一个α螺旋 (19-40位置的氨基酸),共计7个氨基酸的突变导致棘突蛋白RBD和这个突变的ACE2在这个结合位点的结合强度的减弱,具体反映在接触的氨基酸下降到29对(图c)。第二个突变体系只突变了一个氨基酸,也就是重要的K353,变成了His,结果在一个模拟的轨道中平均只有27对氨基酸能够形成紧密接触,并且结合的模式产生了很大变化。这些结果表明这几个突变的氨基酸对于棘突蛋白的结合甚至新冠病毒的入侵起到非常重要的作用。这一研究成果同时搭建了人类ACE2与新冠病毒棘突蛋白RBD复合物的动态结合平台,可以进一步监控及预测新冠病毒棘突蛋白的各种突变(包括可能性)对于复合物结合能力的影响,从而为合理化药物设计及疫苗设计打下坚实的基础。
参考文献:
Computational simulations reveal the binding dynamics between human ACE2 and the receptor binding domain of SARS-CoV-2 spike protein
Cecylia Lupala, Xuanxuan Li, Jian Lei, Hong Chen, Jianxun Qi, Haiguang Liu, XiaodongSu
bioRxiv 2020.03.24.005561; doi: https://doi.org/10.1101/2020.03.24.005561
项目组致谢北京超级云计算中心为抗击新冠肺炎无偿提供的计算资源,以及北京计算科学研究中心天河二号超算集群的主任特批机时。
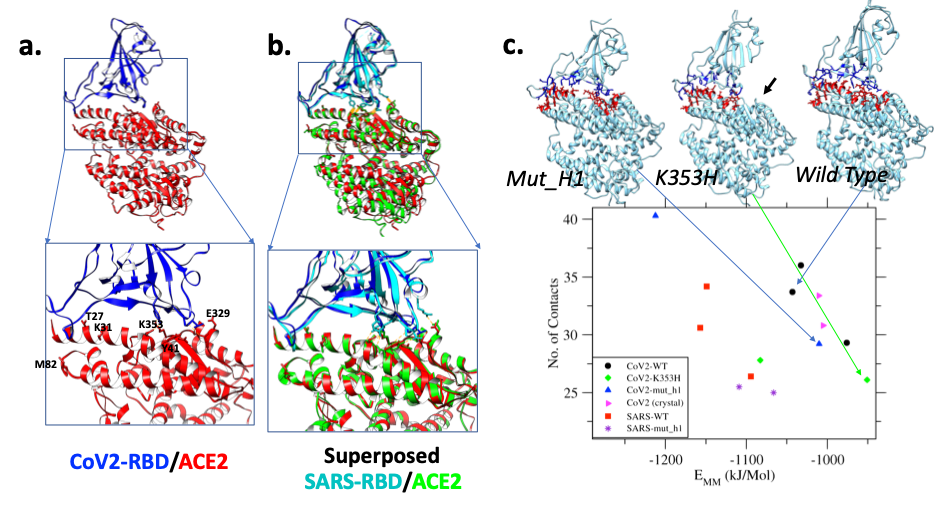
图a,SARS-CoV-2和人类ACE2蛋白复合物的同源建模结构和结合位置附近的放大图;图b,SARS-CoV和人类ACE2复合物的晶体结构的比较;
图c,动力学模拟获得的相互作用信息和对应三种ACE2蛋白的最可能结合模式。与Mut_H1的ACE2结合的棘突蛋白RBD有部分氨基酸脱离了和第一个螺旋的结合;K353H则失去了在第353个氨基酸(从K突变为H)的氢键,以及和最右端第13个ACE2螺旋的相互作用,箭头所示;野生型的ACE2则保持了和初始结构基本一样的结合模式。横轴显示的棘突蛋白与ACE2受体的分子相互作用势能(范德华力和静电势能),真正的结合能需要考虑水解能量和熵的变化对自由能的贡献。



