新冠病毒引发的传染性肺炎(COVID-19)在全球范围传播已经两年了,被世界卫生组织(WHO)列为全球性流行病。新冠病毒因为其球形病毒表面分布着突刺蛋白而形似皇冠,从而得名(即新型冠状病毒,学名SARS-CoV-2)。“皇冠”上的突刺蛋白对于病毒感染细胞至关重要,其途径是通过与人体细胞的血管紧张素转换酶2(简称ACE2)结合,继而与细胞融合之后开始大量复制,进而感染更多的细胞。因此,研究突刺蛋白与ACE2的相互作用的分子机理对于理解新冠病毒致病机理、开发药物切断或者干扰突刺蛋白与ACE2的结合,从而控制病毒感染至关重要。然而,新冠病毒在人群传播过程中会不断发生基因突变,导致对应的蛋白质分子发生变化,所以需要对病毒变种开展持续的监控与研究。最近在南非发现的奥密克戎(Omicron)的变种在突刺蛋白累积了超过30个突变(每个突变代表氨基酸的改变,包括删除、插入、以及替换为其他类型的氨基酸,参见图1)。这个量级的突变给疫苗防护和抗体药物治疗均带来了新的不确定性,而快速传播的Omicron(比野生型和德尔塔的传播性要高很多)也让科学界重新考虑是否有另一条不需要ACE2受体蛋白的病毒入侵途径。
为了解答这些问题,需要尽快分析Omicron新冠变种病毒与ACE2的结合性。北京计算科学研究中心的刘海广课题组和北京大学BIOPIC及生命科学学院的苏晓东实验室合作,应用超级计算机进行了结构建模和动力学模拟,深入系统地分析了Omicron变种的突刺蛋白与人体ACE2的结合模式和动态相互作用细节。分析结果表明Omicron突刺蛋白的受体结合区(Receptor Binding Domain, RBD)与人体ACE2蛋白仍然能够紧密结合,而且RBD本身的稳定性甚至由于Omicron变种携带的突变得到提高。这项成果为理解Omicron的感染机制提供了分子机理,论文已经发表在Biochemical and Biophysical Research Communications(BBRC)。
研究团队基于原始新冠病毒的RBD与ACE2的复合物结构,构造了Omicron变种的结构体系,并通过超级计算机进行优化。整个复合物和水分子模型构成的虚拟溶液体系包含22万个原子。应用分子动力学模拟软件Gromacs,研究人员模拟了500纳秒之内的结构演化。虽然模拟时间相对较短,但是分析结果表明ACE2-RBD复合物已经得到了足够的优化并且收敛到稳定状态。为了降低模拟过程中的随机效应,原始毒株和Omicron的RBD与人体ACE2复合物体系都分别进行了两组独立的动力学模拟。分析发现,Omicron突变体的RBD相比原始毒株的RBD更加稳定,在15个位于RBD区域的突变位点,Omicron变种的RBD都展示了更低的结构扰动,与人体ACE2蛋白形成了新的氢键(比如Q493K和N501Y都引入了新的氢键)和更多的原子接触,并且直接展现在复合物的结合界面面积的增大。研究团队进一步发现Omicron突变导致RBD在于ACE2结和界面的静电势能发生了显著变化,原始毒株的RBD表面零散分布的正电势能区域汇集成更大的区域,从而与ACE2蛋白的静电势能面形成更加完美的互补。这些量化分析的结果与根据蛋白相互作用计算得出的结合自由能数据一致。在全球大流行的背景下,感染人数总量居高不下导致病毒突变越来越多,计算机模型和动力学仿真模拟展示出特有的快速和准确性。在计算模型和结果发表之后,对Omicron变种RBD的预测结果得到了来自加拿大研究团队独立的实验验证。Sriram Subramaniam团队应用冷冻电子显微镜单颗粒成像方法解析出2.66埃分辨率的结构与计算机模拟得出的结果高度一致。
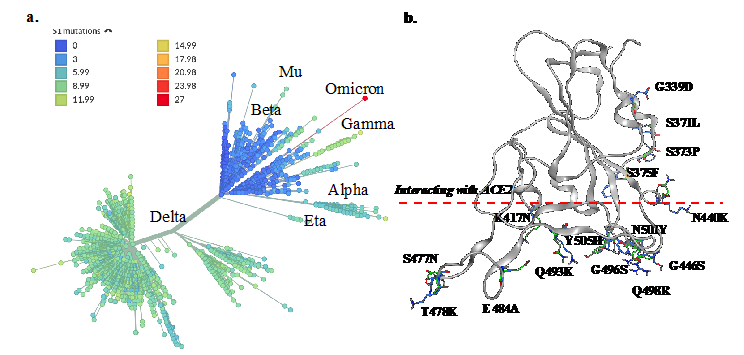
图1. 新冠病毒突变导致的多样性。(a)新冠病毒突变的进化树,其中颜色标度反应突刺蛋白(Spike)上累积的突变个数,Omicron突变显著高于其他的毒株。(b)突刺蛋白的受体结合域RBD氨基酸突变的位置和具体突变种类。

图2.分子动力学模拟得到的代表性结构以及与晶体结构的比较。(a)新冠病毒原始毒株的RBD晶体结构(绿色)与动力学模拟得到的结构对比(原始RBD展示为蓝色,Omicron变种RBD为红色)。(b)三个结构表面的静电势,从红色到蓝色代表的是负电势能到正电势(+/- 5kT/e)。(c)ACE2受体蛋白在于RBD结合界面的静电势能,其中黑色箭头标识的是Omicron变种导致增大的正电势区域和ACE2对应区域的高负电势区域。
致谢 本项目在科技部项目(2021YFC2301300)和中国国家自然科学基金(U1930402, 31971136)的资助下完成,北京计算科学研究中心天河二号超算集群提供了计算机时。
文章链接:Cecylia S. Lupala, Yongjin Ye, Hong Chen, Xiao-Dong Su, Haiguang Liu, Mutations on RBD of SARS-CoV-2 Omicron variant result in stronger binding to human ACE2 receptor, Biochemical and Biophysical Research Communications,
590, 34-41 (2022) https://doi.org/10.1016/j.bbrc.2021.12.079.



